General_Gene_Annotation/
├── CAZy
│ ├── *
│ │ ├── *.CAZy.class.stat.txt [CAZy 数据库注释的结果文件]
│ │ ├── *.CAZy.m8.txt[CAZy 数据库进行 BLAST 比对结果 m8 格式]
│ │ ├── *.CAZy.sumInfo.txt[CAZy 数据库注释的结果文件]
│ │ ├── cazy_summed_up_summary.pdf [CAZy 数据库六大分类统计图,PDF 格式]
│ │ └── cazy_summed_up_summary.png[CAZy 数据库六大分类统计图,PNG 格式]
├── eggnog
│ ├── *
│ │ └── *.emapper.annotations[eggnog结果文件]
├── GO
│ ├── *
│ │ ├── *.gene_go.txt [GO 注释结果按基因汇总]
│ │ ├── *.go.txt[GO 注释结果按分类汇总]
│ │ ├── *.pdf[GO 分类统计图,pdf格式]
│ │ └── *.png[GO 分类统计图]
├── KEGG
│ ├── *
│ │ ├──*kegg.anno.txt 【KEGG 注释结果】
│ │ ├──*.kegg.catalog.txt 【KEGG按照分类汇总基因结果】
│ │ ├──*.kegg.ko.txt【KEGG按照分类汇总KO结果】
│ │ ├──*_map【网页显示KEGG结果】
│ │ │ ├──*KEGG_Map.html【网页图,KEGG通路和KO结果】
│ │ │ ├── base.css
│ │ │ └──* .png
│ │ ├── *.kegg.txt [KEGG 注释结果按分类汇总]
│ │ ├── *.pdf[KEGG 分类统计图,pdf格式]
│ │ └── *.png[KEGG分类统计图]
├── nr
│ ├── *
│ │ ├── *.anno.txt [NR 数据库注释的结果文件]
│ │ ├── *.nr.m8.txt[NR 数据库进行 BLAST 比对结果]
│ │ ├── *.nr.species.anno[NR 数据库物种注释的结果]
│ │ ├── *.nr.species.anno.pdf[NR 数据库物种注释统计图,pdf格式]
│ │ └── *.nr.species.anno.png[NR 数据库物种注释统计图]
├── PFAM
│ ├── *
│ │ ├── *.pfam.gene.tsv [Pfam 注释结果按分类汇总]
│ │ ├── *.pfam.tsv[pfam结果文件]
│ │ └── *.pfam.merge.tsv[Pfam 注释结果按基因汇总]
├── swissProt
│ ├── *
│ │ ├── *.swissProt.m8.txt[Swiss-Prot 数据库进行 BLAST 比对结果]
│ │ └── *.swissProt.mergeInfo.txt[Swiss-Prot 数据库注释的结果文件]
└── TCDB
└── *
├── *.TCDB.anno.tsv[TCDB结果注释文件]
├── tcdb_summed_up_summary.pdf[TCDB 一级分类统计图]
├── tcdb_summed_up_summary.png[TCDB 一级分类统计图]
├── *.TCDB.family.catalog.tsv[TCDB 三级家族分类统计列表]
├── *.TCDB.m8.txt[ TCDB 数据库进行 BLAST 比对结果]
└── *.TCDB.sumInfo.txt[ TCDB 一级分类统计列表]
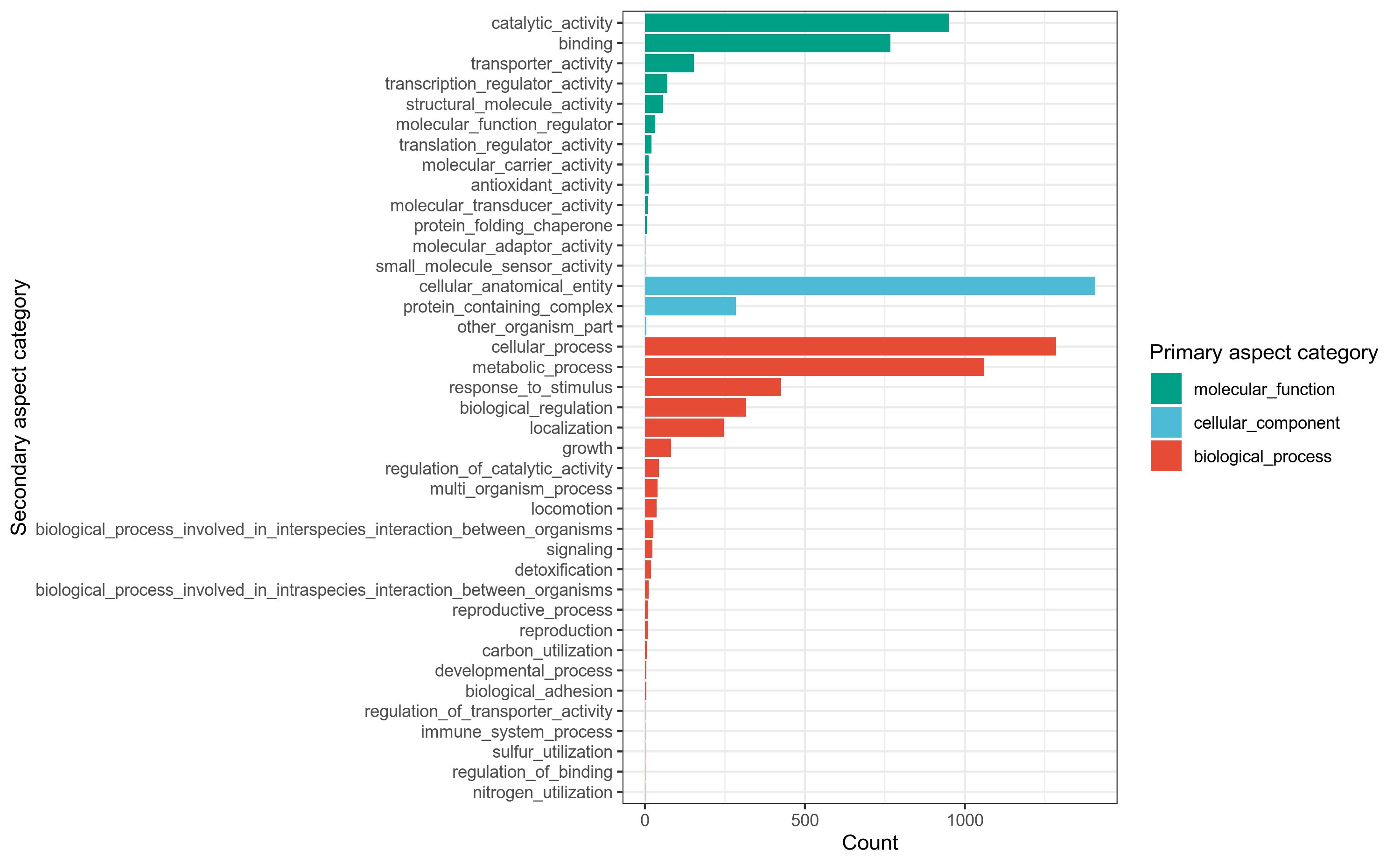
GO(Gene Onotology),是生物学领域公认的,在分子和细胞层面的英文描述词条的参考规范。比如一个蛋白具有某种功能,尽管这是一种具体的功能,但是不同的人可能会有不同的描述,此时如果大家都采用GO里面的规范词条去描述,那么就不会出现很多偏门或者杂乱的描述词汇。GO促进了人们对生物学知识的交流和理解。
GO数据库是基因本体联合会(Gene Onotology Consortium)所建立的数据库,旨在建立一个适用于各种物种的,对基因和蛋白质功能进行限定和描述的,并能随着研究不断深入而更新的语言词汇标准。
GO提供了一系列的词条(terms),用于描绘基因(基因产物)的特点,这些词条分为3大类:
(1) 细胞学组件(cellular component),用于描述亚细胞结构、位置和大分子复合物,例如外部封装结构(external encapsulating structure)等。
(2) 分子功能(molecular function),用于描述基因(基因产物)的功能,比如蛋白质结合转录因子活性(protein binding transcription factor activity)。
(3) 生物学过程(biological process),指的是分子功能的有序组合以实现更复杂的生物功能,例如树突状细胞的抗原处理和递呈(dendritic cell antigen processing and presentation)。
使用emapper[2]注释工具,将细菌基因组prokka注释结果中的.faa文件的基因蛋白序列作为query查询序列,到eggnog蛋白数据库做比对搜索,获得GO注释信息,用R语言做分类统计。
详细结果说明见GO数据库注释结果说明。

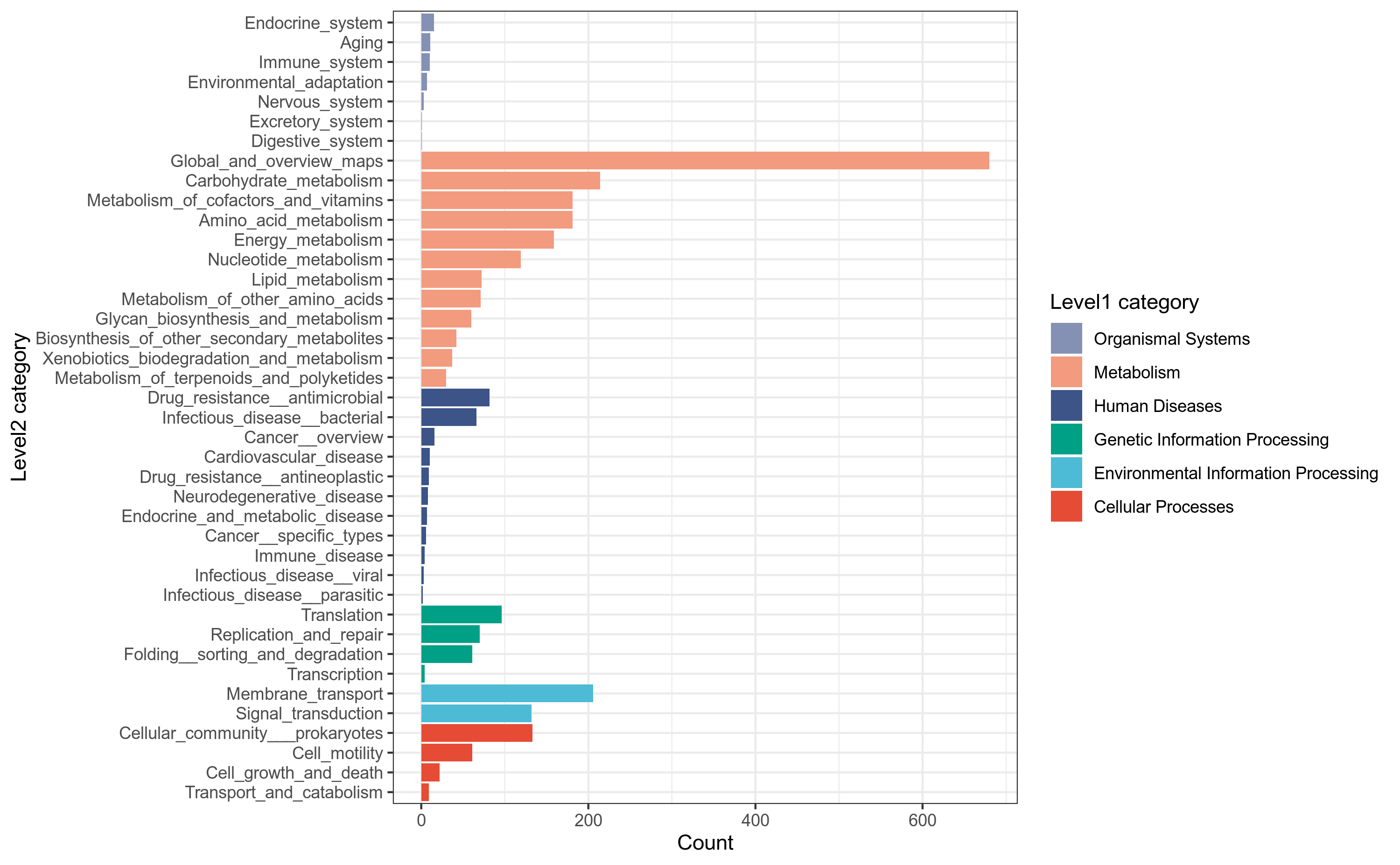
KEGG数据库于 1995 年由 Kanehisa Laboratories 推出 0.1 版,目前发展为一个综合性数据库,其中最核心的为 KEGG PATHWAY 和 KEGG ORTHOLOGY 数据库。在 KEGG ORTHOLOGY 数据库中,将行使相同功能的基因聚在一起,称为 Ortholog Groups (KO entries),每个 KO 包含多个基因信息,并在一至多个 pathway 中发挥作用。
在 KEGG PATHWAY 数据库中,将生物代谢通路划分为以下6类:
(1) 细胞过程(Cellular Processes)
(2) 环境信息处理(Environmental Information Processing)
(3) 遗传信息处理(Genetic Information Processing)
(4) 人类疾病(Human Diseases)
(5) 新陈代谢(Metabolism)
(6) 生物体系统(Organismal Systems)
我们使用emapper注释工具,将细菌基因组prokka注释结果中的.faa文件的基因蛋白序列作为query查询序列,到eggnog蛋白数据库做比对搜索,获得KEGG_Pathway_map_id注释信息,用R语言做分类统计。 详细结果说明见KEGG数据库注释结果说明。
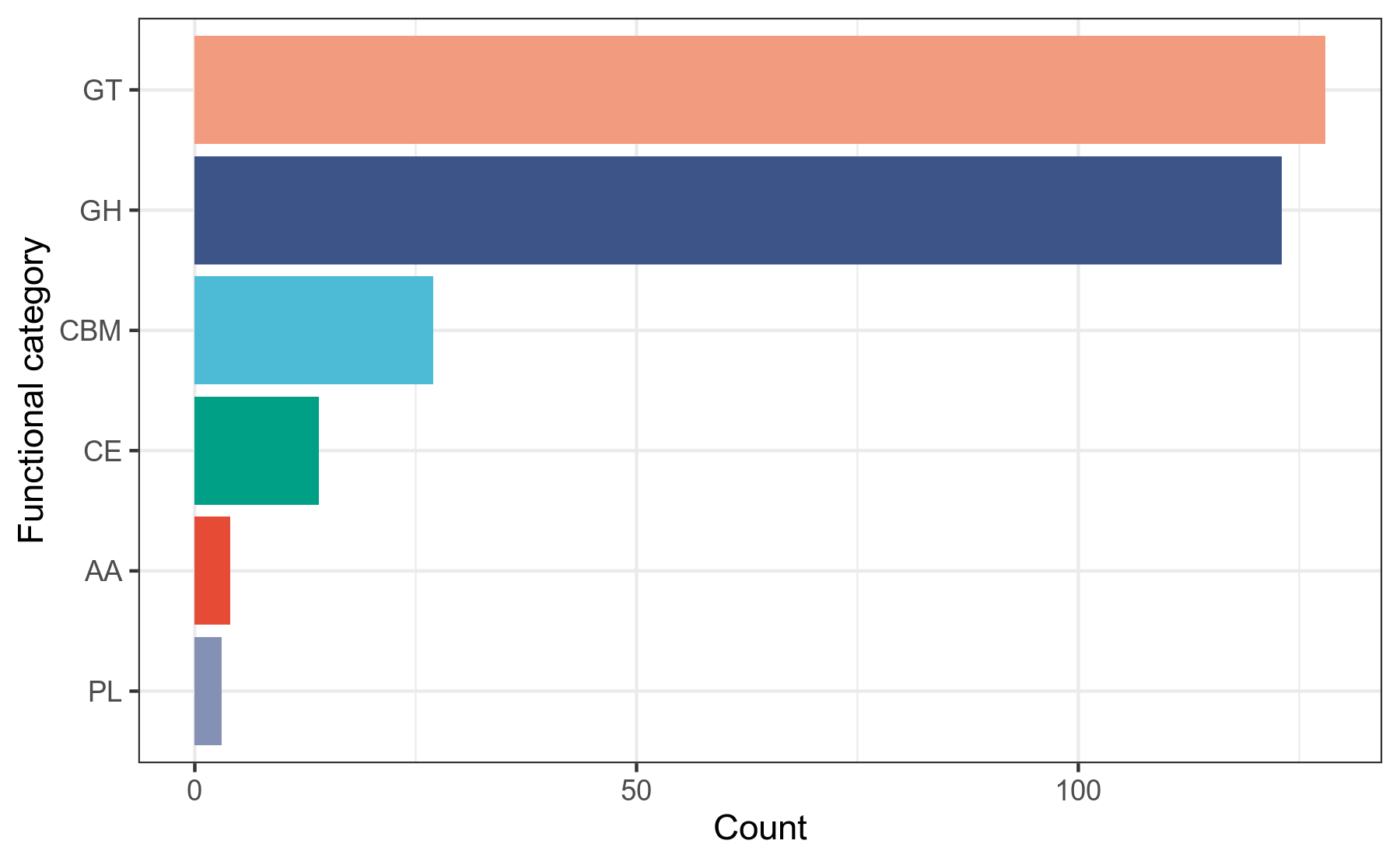
碳水化合物活性酶(CAZy)数据库,录入的是能降解、修饰或者生成糖苷键的酶的功能结构域(或称模块)的信息。
CAZy数据库[4]收录了碳水化合物活性酶的两种常见模块的数据信息:
(1)具有催化活性的模块,分为5类
GH,Glycoside Hydrolases,糖苷水解酶。
GT,Glycosyl Transferases,糖基转移酶。
PL,Polysaccharide Lyases,多糖裂解酶。
CE,Carbohydrate Esterases,碳水化合物酯酶。
AA,Auxiliary Activities,辅助活性模块(一般是氧化还原酶,跟其它的碳水化合物活性酶共同发生作用)。
(2)结合在催化活性模块之上的其它模块,现有1类
CBM,Carbohydrate-Binding Modules,与碳水化合物发生结合作用的模块,一般只起到结合作用,而没有催化作用。
我们使用蛋白序列比对工具diamond,将prokka注释得到的.faa文件中的蛋白序列作为查询序列,到CAZy数据库做比对搜索,获得CAZy数据库注释信息,然后用R语言对注释结果做分类统计。
详细结果说明见CAZy数据库注释结果说明。
SWISS-PROT是经过注释的蛋白质序列数据库,由欧洲生物信息学研究所(EBI)维护。数据库由蛋白质序列条目构成,每个条目包含蛋白质序列、引用文献信息、分类学信息、注释等,注释中包括蛋白质的功能、转录后修饰、特殊位点和区域、二级结构、四级结构、与其它序列的相似性、序列残缺与疾病的关系、序列变异体和冲突等信息。SWISS-PROT中尽可能减少了冗余序列,并与其它30多个数据建立了交叉引用,其中包括核酸序列库、蛋白质序列库和蛋白质结构库等。
我们使用diamond蛋白序列比对工具,将细菌基因组prokka注释结果中的.faa文件的基因蛋白序列作为query查询序列,到Swiss-Prot蛋白数据库做比对搜索,得到细菌的Swiss-Prot蛋白注释信息,生成表格文件。
详细结果说明见SWISS-PROT数据库注释结果说明。
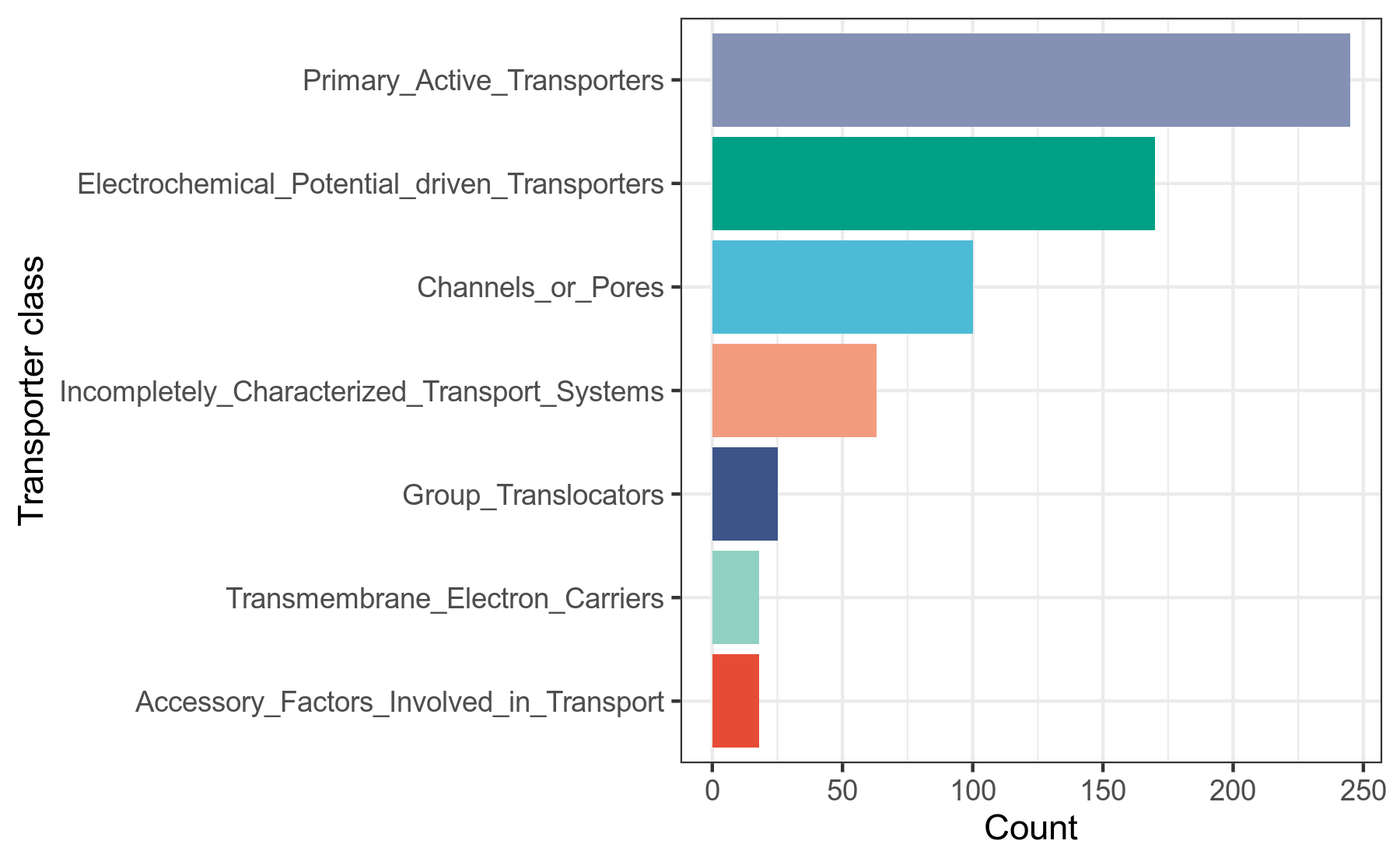
TCDB数据库[5](Transporter Classification DataBase)是对转运蛋白进行分类的一个数据库。类似于对酶进行分类的EC系统,TCDB对于每一个转运蛋白家族,提供了一个TC Nmuber, TC Number 由小数点分隔的5段数字或者字母构成。每一段的数字或字母代表某一个层级的分类,第一级分类包括7个大类。目前TCDB提供了超过800个转运蛋白家族, 包含10000多条唯一的蛋白质序列和10000多篇文献。
我们使用diamond比对工具,将细菌基因组prokka注释结果中的.faa文件的基因蛋白序列作为query查询序列,到TCDB数据库做比对搜索,得到细菌的转运蛋白分类注释信息,然后用R语言做分类统计。 详细结果说明见TCDB数据库注释结果说明。
Pfam数据库[3]可以理解为是Protein family蛋白质家族的英文单词的缩写。该数据库主要提供蛋白质结构域家族的分类信息,被广泛用于查询蛋白质结构域注释信息及其多序列比对信息。在该数据库中,每个蛋白结构域家族由多序列比对和HMMs(hidden Markovmodels,隐马尔可夫模型)所组成。Pfam-A根据最新的UniProtKB蛋白序列数据库所构建而成,是人工注释和检查的蛋白结构域信息数据库,可信度较高。pfam_scan是Pfam官网提供的工具软件,用来分析蛋白序列具有哪些结构域。
我们使用pfam_scan分析工具,以Pfam-A数据库作为参考数据库,对细菌基因组prokka注释结果中的.faa文件的基因蛋白序列做注释,得到蛋白质结构域家族注释信息,生成表格文件。
详细结果说明见PFAM数据库注释结果说明。
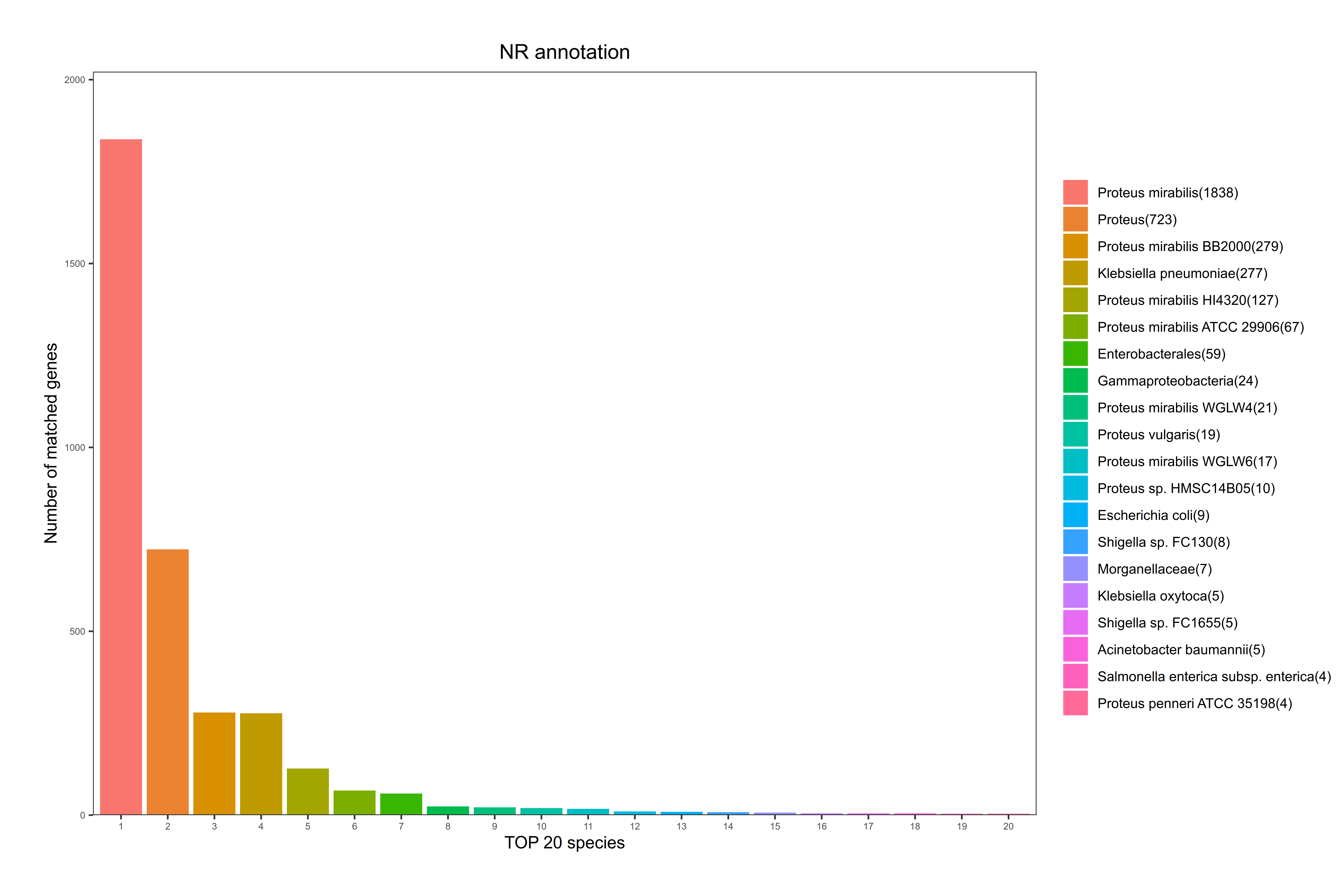
NR全称为Non-Redundant Protein Database,是一个非冗余的蛋白质数据库,由NCBI创建并维护,其特点在于内容比较全面,同时注释结果中会包含有物种信息,可作物种分类用。
详细结果说明见nr数据库注释结果说明。
根据基因注释到的物种情况,统计注释到的物种及基因数目,其统计结果如下图:
| 软件 | 版本 |
|---|---|
| Diamond | v2.0.14 |
| eggNOGmapper | 104.3 |
| pfam | 35.0 |
| CAZy | 2022 |
| tcdb | 2022 |
| emapper | 2.0.1 |