温馨提示:请使用火狐或者Chrome的网页浏览器来查看报告
微科盟宏基因组分箱数据分析结题报告
一 概述
宏基因组分箱(Binning)是将宏基因组测序得到的混合了不同微生物的序列reads或序列组装得到的contigs或scaffolds按物种分开归类的过程。这些分开归类的序列被称为宏基因组组装基因组(metagenome-assembled genomes,MAGs)。传统的单物种全基因组序列都是经纯培养之后,再进行全基因组de novo测序才获得的,但是环境中存在着大量的不可培养微生物(uncultured candidate bacterial species,未培养候选菌种),而宏基因组分箱及相关技术不仅有助于获得不可培养微生物的基因组序列,还有以下诸多功能:(1)发现新物种,预测新物种基因,利用现有数据库分析新物种的功能;(2)扩充微生物基因组数据库,增加微生物多样性,提高宏基因组数据reads mapping率(检出率);(3)有助于宏基因组技术开发;(4)助力“感兴趣”微生物群结构和功能的研究;(5)为菌群的分类和功能描述提供了更多的解决方案。早在2011年,science上的一篇文章就用了宏基因组Binning技术对来自牛瘤胃的样本进行了宏基因组测序研究。该研究从268 Gbp的宏基因数据中成功Binning出了15个不能培养的微生物的全基因组序列[1]。
从那以后,宏基因组Binning技术开始被更多的人关注和重视,也逐渐出现了很多宏基因组Binning相关的工具。Metabat是近几年所有分箱工具中最受欢迎的工具(引用达460+)。2019年发表在PeerJ上的新版Metabat2更是在完成度、效率等多方面均优于Metabat和同类工具 [2-3]。仅在2019年当年Metabat2就已经被Cell、Nature Biotechnology、Genome Biology等多篇高水平期刊引用 [4-6]。因此微科盟从众多分箱工具中选择Metabat2进行分箱。不仅如此,微科盟宏基因组分箱分析流程种使用的每一个软件都是根据效率、准确度等多个参数从众多同类软件中精挑出来的,例如序列组装工具Megahit,Bin质检工具Checkm,功能取预测工具Prokka,蛋白比对工具Diamond等等。
二 项目流程
2.1 实验流程
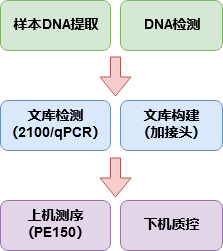
图2-1 测序流程图
2.2 数据分析流程
如图2-2,宏基因组分箱的数据分析流程如下:
- 数据质控:测序得到的原始数据会存在一定比例的低质量数据,为了保证后续信息分析结果的准确可靠,首先要对原始数据进行质控及宿主过滤,得到有效数据[7]。分析中将使用Kneaddata软件彻底清除原始数据中的Illumina接头序列、低质量的序列片段和较短序列。质控前和质控后,会用FastQC来检测质控的合理性和效果。
- 去除宿主:质控处理后的数据通过Bowtie2软件[9]比对到宿主的基因组(整合到了Kneaddata软件里),没有比对到的序列被保留下来做后续分析。
- 分箱:对于每个样本,运用Megahit软件,将样本去宿主基因后的clean reads进行组装(megahit默认组装参数),得到contigs;运用Bowtie2进行建索引和序列比对获得clean reads在contigs中的比对信息;用Samtools软件[10]对比对结果进行格式转换和排序;用Metabat2自带的jgi_summarize_bam_contig_depths程序计算contigs深度;挑选长度大于1500bp的contigs,结合上一步得到的contigs深度,用Metabat2对contigs进行分箱;用RefineM [11] 对得到的bins进行提纯,去除bins中污染度较高的contigs
- 去冗余和筛选:合并所有样本中得到的bins,用CheckM[12]中的lineage_wf流程评估bins的完成度和污染度,并用dRep[13]软件对bins进行筛选和去冗余,默认污染度筛选阈值50%,完整度筛选阈值50%(不固定,分析前请和技术支持沟通确认筛选阈值)。
- Bin可视化和丰度分析:首先计算每个Bin中每条序列的GC含量,结合分箱过程中得到的contig depth数据进行Bin可视化,根据contig的GC含量和depth信息绘制二维散点图;在Metawrap环境中,使用Metawrap的quant_bins模块(Salmon算法)计算每个Bin的丰度。如果每组样品数大于等于3,接着用lefse进行差异丰度分析寻找组间差异Bin。
- Bin功能注释:使用Prokka软件[15]对每个Bin进行功能注释,获得每个Bin中的rRNA、tRNA、tmRNA、基因、直系同源蛋白簇(COG)、EC注释信息。使用KofamKOALA[16]的离线版本Kofamscan进行KEGG功能注释。使用eggnog-mapper[17]进行GO注释。使用Diamond软件[18]比对CAZy数据库对每个Bin进行碳水化合物酶注释,获取每个Bin中的碳水化合物酶信息。
- Bin物种注释与进化分析:使用 PhyloPhlAn3软件[19] 将bins与SGB release of September 2020 [4]比对,获取每个bin的物种分类信息;并基于Prokka得到的蛋白序列信息,计算bins之间的进化树
- Bin圈图分析:利用每个Bin(基于contig)的Prokka蛋白预测信息,功能区注释信息(含正负链、CDS、RNA类型等信息),CAZy数据库注释结果(GH、GT、CE、PL、CBM、AA),以及GC content、GC skew的统计结果绘制Circos圈图,直观展示整个Bin的功能注释信息。

图2-2 宏基因组分箱数据分析流程
三 结果文件夹解读
00-QCStats序列质控和去宿主序列
|--00-QCStats | |--1-QC_report_Rawfastq/*.html [原始数据fastqc质检结果] | |--2-QC_report_Filtered/*.html [序列质控和去宿主序列之后的fastqc质检结果] | |--reads_summary.txt [数据产出质量情况一览表]
本项目采用Illumina Novaseq平台对测序样本进行双端测序。基于FASTQ格式的测序文件是一种存储序列信息的特定文件,推荐用Notepad++等文本编辑器或者在电脑终端中打开。FASTQ文件每四行对应一条测序Read:第一行以符号“@”起始,对应于序列ID和相应的描述信息;第二行为实际测得的碱基序列;第三行以符号“+”起始;而第四行的字符串则记录了第二行序列中每个碱基所对应的测序质量(详见 https://en.wikipedia.org/wiki/Fastq)。
采用 Illumina测序平台测序获得的原始数据(Raw Data)存在一定比例低质量数据,为了保证后续分析的结果准确可靠,需要对原始的测序数据进行预处理,包括质控(Trimmomatic[20] 参数:ILLUMINACLIP:adapters_path:2:30:10 SLIDINGWINDOW:4:20 MINLEN:50),和去宿主序列(Bowtie2[9]参数:--very-sensitive),获取用于后续分析的有效序列(clean data)。测序数据预处理统计结果见表 3-1。序列质控步骤关键参数解释如下:
1) 去除接头序列 (参数ILLUMINACLIP:adapters_path:2:30:10);
2) 扫描序列(4bp滑窗大小),如果平均质量分低于20(99%正确率),切除后续序列(参数SLIDINGWINDOW:4:20);
3) 去除最终长度小于50bp的序列(参数MINLEN:50)。
| Sample ID | InsertSize(bp) | SeqStrategy | RawReads(#) | Raw Base(GB) | %GC | Raw Q20(%) | Raw Q30(%) | Clean Reads(#) | Cleaned(%) | Clean Q20(%) | Clean Q30(%) |
|---|---|---|---|---|---|---|---|---|---|---|---|
| C1 | 350 | (150:150) | 23613034 | 7.08 | 61 | 97.70 | 93.78 | 22416119 | 94.93 | 98.80 | 95.62 |
| C2 | 350 | (150:150) | 27584954 | 8.28 | 61 | 97.10 | 92.62 | 26060963 | 94.48 | 98.55 | 94.94 |
| C3 | 350 | (150:150) | 23777022 | 7.13 | 61 | 98.45 | 95.71 | 22889304 | 96.27 | 99.27 | 97.08 |
| T11 | 350 | (150:150) | 29809022 | 8.94 | 62 | 97.62 | 93.48 | 28346006 | 95.09 | 98.63 | 95.16 |
| T12 | 350 | (150:150) | 24702558 | 7.41 | 61 | 97.72 | 93.78 | 23570823 | 95.42 | 98.72 | 95.43 |
| T13 | 350 | (150:150) | 28274974 | 8.48 | 61 | 97.63 | 93.54 | 26703496 | 94.44 | 98.69 | 95.32 |
| C4 | 350 | (150:150) | 27007788 | 8.10 | 61 | 97.63 | 93.65 | 25636029 | 94.92 | 98.77 | 95.52 |
| T21 | 350 | (150:150) | 25016746 | 7.51 | 60 | 97.74 | 93.74 | 23831131 | 95.26 | 98.70 | 95.35 |
| T22 | 350 | (150:150) | 24980036 | 7.49 | 60 | 97.55 | 93.21 | 23555813 | 94.30 | 98.57 | 94.96 |
| T23 | 350 | (150:150) | 24026221 | 7.21 | 62 | 97.64 | 93.58 | 22758509 | 94.72 | 98.69 | 95.34 |
表3-1 数据产出质量情况一览表
01-Bin分箱与bins质控
文件说明: |--01-Bin | |--1-Bin_raw/*.fa [所有样本分箱得到的bins] | |--2-Bin_pick/*.fa [去冗余,筛选后的bins] | |--bin_picked_summary.txt [统计所有筛选得到的bins:完成度、污染度、N50、N90、Size、GC含量] | |--checkm_raw.txt [所有bins的污染度、完整度]
对每个样本,使用Megahit组装Clean Reads测序数据,得到contigs。分别用Bowtie2和Samtools进行比对和格式转换。得到conitgs深度数据后,使用Metabat2进行宏基因组分箱。设置Metabat2参数“-m 1500”,即使用1500bp以上的conitgs进行分箱; 用RefineM [11] 对得到的bins进行提纯,去除bins中污染度较高的contigs。使用Checkm对每个bin进行质量检查,获得原始bins的完成度和污染度信息。并用dRep软件对bins进行筛选和去冗余,默认污染度筛选阈值50%,完整度筛选阈值50%(不固定,分析前请和技术支持沟通确认筛选阈值)。
| BinID | Completeness | Contamination | N50 | N90 | Size | GC_percent |
|---|---|---|---|---|---|---|
| bin.C2.4 | 83.18 | 3.24 | 5779.0 | 2407.0 | 2757464.0 | 0.519731 |
| bin.C3.3 | 94.39 | 2.56 | 17120.0 | 5802.0 | 2387822.0 | 0.486806 |
| bin.T11.34 | 84.77 | 1.95 | 11213.0 | 4482.0 | 2172812.0 | 0.656988 |
| bin.T11.39 | 84.97 | 8.48 | 7877.0 | 2804.0 | 4755645.0 | 0.704336 |
| bin.T11.40 | 83.81 | 2.14 | 13994.0 | 4941.0 | 3526782.0 | 0.390611 |
| bin.T11.43 | 89.18 | 3.66 | 11872.0 | 4352.0 | 5668033.0 | 0.664217 |
| bin.T11.49 | 91.87 | 5.06 | 18033.0 | 4949.0 | 2858976.0 | 0.383393 |
| bin.T11.51 | 83.05 | 1.48 | 12386.0 | 4727.0 | 2538282.0 | 0.429579 |
| bin.T11.53 | 82.83 | 8.60 | 6716.0 | 2609.0 | 5816971.0 | 0.661194 |
| bin.T11.56 | 80.63 | 0.99 | 14305.0 | 5869.0 | 4221543.0 | 0.496679 |
| bin.T11.62 | 95.01 | 2.70 | 26802.0 | 9418.0 | 3869024.0 | 0.681719 |
| bin.T11.63 | 91.50 | 7.19 | 17177.0 | 5403.0 | 6242153.0 | 0.626916 |
| bin.T11.8 | 85.82 | 1.60 | 42723.0 | 16080.0 | 2157794.0 | 0.441075 |
| bin.T12.1 | 92.75 | 4.03 | 21627.0 | 6477.0 | 2668968.0 | 0.696741 |
| bin.T12.2 | 88.25 | 12.02 | 7572.0 | 2754.0 | 4771274.0 | 0.704780 |
| bin.T12.40 | 87.86 | 0.48 | 52664.0 | 18224.0 | 3137211.0 | 0.459016 |
| bin.T12.45 | 95.22 | 0.00 | 103547.0 | 19864.0 | 6096027.0 | 0.514886 |
| bin.T13.21 | 84.74 | 3.45 | 11091.0 | 3827.0 | 2956239.0 | 0.429620 |
| bin.T13.23 | 91.47 | 3.96 | 11925.0 | 4164.0 | 3312802.0 | 0.727346 |
| bin.T13.2 | 82.01 | 6.02 | 7608.0 | 2792.0 | 4338635.0 | 0.592594 |
| bin.T13.32 | 86.60 | 3.26 | 12397.0 | 3885.0 | 3699007.0 | 0.622773 |
| bin.T13.35 | 90.75 | 11.76 | 8825.0 | 3108.0 | 4771640.0 | 0.704238 |
| bin.T13.38 | 93.07 | 1.69 | 18536.0 | 6312.0 | 2319053.0 | 0.446278 |
| bin.T13.3 | 90.59 | 1.57 | 22386.0 | 6338.0 | 4174077.0 | 0.597061 |
| bin.T13.48 | 94.55 | 2.75 | 16538.0 | 4890.0 | 3356734.0 | 0.490223 |
| bin.T13.53 | 94.41 | 2.87 | 14408.0 | 4450.0 | 2508548.0 | 0.402786 |
| bin.T13.54 | 92.72 | 8.48 | 24943.0 | 8688.0 | 4219818.0 | 0.671570 |
| bin.T13.55 | 87.78 | 4.86 | 8588.0 | 3215.0 | 3041405.0 | 0.369481 |
| bin.T13.56 | 98.62 | 0.73 | 58745.0 | 17030.0 | 3953903.0 | 0.422191 |
| bin.T13.57 | 92.66 | 10.43 | 11662.0 | 3593.0 | 3496934.0 | 0.691131 |
| bin.T13.58 | 97.16 | 3.08 | 34919.0 | 10446.0 | 6324461.0 | 0.611210 |
| bin.T13.7 | 99.51 | 1.23 | 63466.0 | 22628.0 | 3274520.0 | 0.372735 |
| bin.T13.8 | 85.41 | 1.97 | 9259.0 | 3455.0 | 3069200.0 | 0.426120 |
表3-2 筛选得到的bins统计
02-Bin_Plot 分箱效果散点图
文件说明: |--02-Bin_Plot | |--bins_contig_summary.txt [散点图输入文件,含每个Bin的contig的GC含量和contig的深度信息] | |--bins_contig_summary.pdf [绘图结果,利用GC和depth数据把每个bin的contig 画到图中]
利用可视化的方法直观展示Bin的由来和所含信息。宏基因组分箱的原理是根据序列(contig/scaffold)四核苷酸频率和序列丰度变化模式将序列分成一个个bins。每个bin的每个contig都会在此处理(metabat2分箱)的过程中有一个depth深度数据。另外我们利用自己的python脚本计算每个bin的contig GC含量。有了contig GC含量和depth数据即可进行Bin可视化,绘制每个Bin中每个contig的散点图(图3-1)。
图3-1 Binned contigs可视化
横坐标是contig的GC含量;纵坐标是contig depth;一个点代表一个contig,相同颜色的contig来自同一个bin。
03-Bin_Abundance 丰度分析
03-Bin_Abundance ├── 1-Barplots [丰度柱形图] ├── 2-Heatmaps [丰度热图] ├── 3-Circos [丰度圈图] ├── 4-SignificanceAnalysis [丰度组间显著性差异比较] ├── 5-CorrelationAnalysis [丰度相关性分析] └── bin_abundance_table.xls [bins在各样本的丰度汇总表]
首先,用metawrap中的quant_bins模块(salmon算法),估计每个样品中每个scaffold的丰度,计算Bin平均丰度,获得样品Bin丰度表。Bin丰度是“每百万序列基因组拷贝数”,是已经标准化的数据,类似RNAseq分析中的TPM(每百万转录本)。接着,用R语言pheatmap绘图函数绘制样品-Bin丰度热图。热图可以以色块颜色深浅的方式表达Bin丰度的大小,还可以进行Bin-Bin聚类和样品-样品聚类,相似的丰度模式会被聚到一起。然后,统计每个Bin的丰度总数,用ggplot geom_bar绘制柱形图并排序,展示整批数据中所有Bin的丰度情况。如果客户提供的样品分组>=2且每组样品在三个以上,那么还可以用lefse进行组间差异分析寻找与分组有关的Bin。
1-Barplots文件夹中包含按照分组顺序排列的每个样本的bin丰度柱形图(图3-2),以及计算分组均值后画的柱形图。
图3-2 丰度柱形图
横坐标(Sample Name)是样品名,纵坐标(Sequence Number Percent)表示bin的丰度占总丰度的比率,柱状图自上而下的颜色顺序对应于右侧的图例颜色顺序。图例中最多显示最优势的20个bins,余下的相对丰度较低的bin被归类为Other在图中展示。
2-Heatmaps文件夹中包含按照分组顺序排列的每个样本的bin丰度热图(图3-3),以及计算分组均值后画的热图。
图3-3 丰度聚类热图
说明:纵轴为样品名称信息,同时也包括了分组信息。横轴为bin ID。图中上方的聚类树为bin在各样本中丰度分布的相似度聚类,左侧的聚类树为样品聚类树,中间的热图是bins的相对丰度(log10(TPM))热图,颜色与相对丰度的关系见图上方的刻度尺。
3-Circos 文件夹中包含按照分组顺序排列的每个样本的bin丰度Circos图(图3-4),用于展示每个样本各个bin(丰度前10)的丰度比例,以及每个bin在各个样本中的丰度比例。
图3-4 丰度Circos图
说明:左半圈为丰度最高的十个bin,每个bin内,不同颜色代表不同样本来源的丰度比例;右边半圈为样本,每个样本内不同颜色代表不同bin的丰度比例。
4-SignificanceAnalysis文件夹中,DunnTest文件夹包含了DunnTest组间多重比较结果表格,第一列为bin ID, KW_pvalue是Kruskal-Wallis算出总的p值,DunnTest_comparison是比较的分组对,DunnTest_Z是DunnTest检验统计量,DunnTest_PValueAdjusted是Bofferoni校正错误发现率后的DunnTestp值;LEfSe文件夹中包含了LEfSe分析中,LDA分值大于2的bins柱形图(图3-5),LEfSe寻找每一个分组的特征bins(默认为LDA>2的bin)[21],也就是相对于其他分组,在这个组中丰度较高的bin。
图3-5 LEfSe分析LDA柱形图
说明:每一横向柱形体代表一个bin,柱形体的长度对应LDA值,LDA值越高则分组间该bin的丰度差异越大。柱形的颜色对应该bin是那个分组的特征bin(在对应分组中的丰度相对较高)。
只有提供了环境因子,5-CorrelationAnalysis中才会包含内容。其中,CorrelationHeatmap文件夹中包含了相关性热图(图3-6);RDA文件夹中包含了RDA分析结果图(图3-7)。CCA/RDA的分析主要依赖R语言VEGAN包,以及用ggplot2进行可视化。CCA/RDA(DCA判断用哪一种分析)分析是基于对应分析发展的一种排序方法,将对应分析与多元回归分析相结合,每一步计算均与环境因子进行回归,又称多元直接梯度分析。RDA是基于线性模型,CCA是基于单峰模型。本报告先进行DCA分析,看最大轴的值是否大于4,如果大于4.0,就选CCA,否则选RDA。该分析主要用来反映菌群与环境因子之间的关系,可以检测环境因子、样品、菌群(抗性基因,KEGG功能)三者之间的关系或者两两之间的关系,可得到影响样品分布的重要环境驱动因子。该分析给出的所有p值都是反映解释变量(连续的数值变量,或者分类变量)对bins群落丰度结构的解释程度是否显著(简单的说就是解释变量对bins 丰度是否有影响,影响是否显著),所有p值都是用R语言VEGAN包里的置换检验得出的(permutation test),*_features_location_plot图中(图3-7)的p值反映了所有连续的数值变量(环境因子)对bins丰度差异的解释程度(总的p值),表格*_RDA.envfit中的p值反映了每个环境因子对bins丰度差异的解释程度;*_RDA_sample_location_plot图中的p值反映了分组对bins丰度差异的解释程度,p<0.05,解释方差显著,图中样本点之间的距离近似于样本之间bins丰度结构差异程度,样本点投影到环境因子对应的值近似于该样本真实的环境因子值。
图3-6 bins与环境因子之间的相互关系热图
说明:X轴上为环境因子,Y轴为bins。计算获得R值(秩相关)和校正错误发现率的P值。R值在图中以不同颜色展示,右侧图例是不同R值的颜色区间。* 0.01≤ P <0.05,** 0.001≤P < 0.01,*** P < 0.001。
图3-7 bins CCA/RDA排序图
说明:在CCA/RDA bins排序图内,环境因子用箭头表示,箭头连线的长度代表某个环境因子与bins丰度分布间相关程度的大小(解释方差的大小),箭头越长,说明相关性越大,反之越小。箭头连线和排序轴的夹角代表某个环境因子与排序轴的相关性大小,夹角越小,相关性越高;反之越低。环境因子之间的夹角为锐角时表示两个环境因子之间呈正相关关系,钝角时呈负相关关系。每个点代表一个bins,点越大,对应bins丰度越高,灰色点代表丰度较低的bins,未在图中注释bins名称,将bins投影到各个环境因子,对应的值即为该bins倾向于存在的环境(喜欢的环境)。或者说,将bins点与原点连线,bins间,bins与环境因子间,环境因子间的夹角的余弦值近似于相关系数,至于有多近似,就要看RDA1/CCA1和RDA2/CCA2两个坐标轴的解释方差百分比有多大,越大越近似。
04-Bin_Function bins功能注释情况统计
04-Bin_Function [功能注释结果文件夹] ├── CAZyme [CAZy碳水化合物数据库注释结果] ├── COG [eggNOG数据库COG注释结果] ├── GO [GO数据库注释结果] ├── KEGG [KEGG数据库注释结果] └── * ├── *_detail_bin [包含每个bin 注释到数据库基因家族的种类和CDS个数统计图] | └── *_annotations.txt [每个bin的原始注释结果文件] ├── *_detail_bin_L1 [包含每个bin 注释到数据库基因家族level1水平的种类和CDS个数统计图] ├── *_detail_gene [包含每个注释到的数据库基因家族来源于各个bin 的CDS个数统计图] ├── *_summary [注释情况汇总统计图,由于图片大小限制,只会展示CDS个数排名靠前的基因家族和bins] ├── *_GeneCount.xls [bins注释到数据库基因家族的CDS个数统计表] └── *_GeneCount.L1.xls [bins注释到数据库基因家族level1水平的CDS个数统计表]
04-Bin_Function文件夹包含了各个功能数据库的注释情况统计;接下来我们将对每个功能数据库展开说明:
04-Bin_Function/CAZyme CAZy碳水化合物数据库注释
碳水化合物酶(CAZy)数据库是关于能够合成或者分解复杂碳水化合物和糖复合物的酶类的数据库。CAZy数据库基于蛋白质结构域中的氨基酸序列相似性将碳水化合物活性酶类归入不同蛋白质家族。CAZy数据库提供了酶分子序列的家族信息,物种来源,基因序列,蛋白序列,三维结构,EC分类,相关数据库链接。此数据库可以将酶分子的序列、结构、催化机制关联起来。CAZy有三个level,第一个level是六大功能类,即GH GT AA CE PL CBM;第二个level是CAZy family;第三个是有EC编号的具体酶信息,如EC2.4.1.129。
利用蛋白比对工具diamond将prokka预测得到的所有功能区(CDS)与CAZy数据库进行比对可获取基因组CAZyme family注释信息。
碳水化合物活性酶数据库中CAZymes功能分类如下:
| 名称 | 缩写 |
|---|---|
| 糖苷水解酶类 | GHs |
| 糖苷转移酶类 | GTs |
| 多糖裂解酶类 | PLs |
| 糖水化合物脂酶类 | CEs |
| 碳水化合物结合模块 | CBMs |
| 辅助模块酶类 | AAs |
部分结果统计图如下:
图3-8 Level1 CAZymes数量统计箱图
说明:横坐标是 CAZymes的level1大类,纵坐标是每一类CAZyme在各个bin中的CDS数量,每个箱子的高度表示CAZy数量的变化幅度,箱子的首和尾是极值,中间的横线是中位数。
图3-9 Level1 CAZymes数量统计热图
说明:pheatmap热图可以展示每个Bin的CAZyme CDS计数,不同Bin之间的相似度(横向)和不同CAZyme类别之间的相似度(纵向)。图中每一个色块表示一个Bin中的一类CAZyme CDS计数,颜色越蓝表示个数越低,越红表示个数越高。
图3-10 Level1 CAZymes数量统计堆叠图
说明:一种颜色表示一类CAZyme;色柱的高度表示该CAZy的CDS计数
图3-11 Level1 CAZymes数量统计饼图
说明:该饼图展示每个bin中的每一个CAZyme类别CDS计数与该bin中总数量的比例。
图3-12 某个CAZyme 在每个bin中的CDS数量统计饼图
说明:图片篇幅限制,只展示CDS数量排名前10的bin
04-Bin_Function/COG COGs注释
COG,即Clusters of Orthologous Groups of proteins(同源蛋白簇)。COG是由NCBI创建并维护的蛋白数据库,根据细菌、藻类和真核生物完整基因组的编码蛋白系统进化关系分类构建而成。COG分为两类,一类是原核生物的(一般称COG),另一类是真核生物(一般称KOG)。通过比对可以将某个蛋白序列注释到某一个COG中,每一簇COG由直系同源序列构成,从而可以推测该序列的功能。
图3-13 展示了COG level2水平其中一个bin的CDS计数统计柱形图,其它bins请查看结果文件夹。
图3-13 COG数据库分类注释统计图
说明:该图描述的是预测基因(CDS)注释到COG不同分类水平数量情况;纵坐标是COG level 2分类(字母表示,共26种分类);横坐标是注释到的CDS计数;颜色是COG level 1分类(四大类,分别是:细胞过程和信号传递、信息储存和加工、代谢和尚未明确);柱子越长,属于该分类的预测基因越多。
COG level 2分类的26个字母的意义如下:
| COG Function Category | Level 1 | Level 2 |
|---|---|---|
| J | INFORMATION STORAGE AND PROCESSING | Translation, ribosomal structure and biogenesis |
| A | INFORMATION STORAGE AND PROCESSING | RNA processing and modification |
| K | INFORMATION STORAGE AND PROCESSING | Transcription |
| L | INFORMATION STORAGE AND PROCESSING | Replication, recombination and repair |
| B | INFORMATION STORAGE AND PROCESSING | Chromatin structure and dynamics |
| D | CELLULAR PROCESSES AND SIGNALING | Cell cycle control, cell division, chromosome partitioning |
| Y | CELLULAR PROCESSES AND SIGNALING | Nuclear structure |
| V | CELLULAR PROCESSES AND SIGNALING | Defense mechanisms |
| T | CELLULAR PROCESSES AND SIGNALING | Signal transduction mechanisms |
| M | CELLULAR PROCESSES AND SIGNALING | Cell wall/membrane/envelope biogenesis |
| N | CELLULAR PROCESSES AND SIGNALING | Cell motility |
| Z | CELLULAR PROCESSES AND SIGNALING | Cytoskeleton |
| W | CELLULAR PROCESSES AND SIGNALING | Extracellular structures |
| U | CELLULAR PROCESSES AND SIGNALING | Intracellular trafficking, secretion, and vesicular transport |
| O | CELLULAR PROCESSES AND SIGNALING | Posttranslational modification, protein turnover, chaperones |
| X | CELLULAR PROCESSES AND SIGNALING | Mobilome: prophages, transposons |
| C | METABOLISM | Energy production and conversion |
| G | METABOLISM | Carbohydrate transport and metabolism |
| E | METABOLISM | Amino acid transport and metabolism |
| F | METABOLISM | Nucleotide transport and metabolism |
| H | METABOLISM | Coenzyme transport and metabolism |
| I | METABOLISM | Lipid transport and metabolism |
| P | METABOLISM | Inorganic ion transport and metabolism |
| Q | METABOLISM | Secondary metabolites biosynthesis, transport and catabolism |
| R | POORLY CHARACTERIZED | General function prediction only |
| S | POORLY CHARACTERIZED | Function unknown |
04-Bin_Function/GO GO注释
GO数据库是基因本体联合会(Gene Onotology Consortium)所建立的数据库,旨在建立一个适用于各种物种的,对基因和蛋白质功能进行限定和描述的,并能随着研究不断深入而更新的语言词汇标准。GO是多种生物本体语言中的一种,是OBO(Open BiomedicalOntologies)组织中的一员,GO提供了一系列的语义(terms)用于描绘基因、基因产物的特点,这些语义通过三个概念维度展开:细胞学组件(Cellular Component)用于描述某个节点的亚细胞结构、位置和大分子复合物,如外部封装结构(external encapsulating structure)等;分子功能(molecular function),用于描述基因以及基因产物的功能,比如蛋白质结合转录因子活性(protein binding transcription factor activity);生物学途径(biological process)指的是分子功能的有序组合以实现更复杂的生物功能,例如树突状细胞的抗原处理和提呈(dendritic cell antigen processing and presentation)。
使用eggnog-mapper功能注释软件和eggNOG数据库进行GO注释。该方法除了获得GO的注释信息还能获得KEGG、COG等信息。获得GO结果后统计注释到每个GO中的基因数量,然后进一步归类获得GO的category分类信息。最后通过R语言对该数据进行柱形图绘制。
图3-14 展示了其中一个bin预测基因所属GO的计数,其它bins请查看结果文件夹。
图3-14 GO注释统计图
该图说明的是其中一个bin预测基因所属GO的情况;横坐标是数量;纵坐标是GO;颜色是GO分类;柱子越高,该GO中包含的预测基因(CDS)越多。
04-Bin_Function/KEGG KEGG注释
KEGG数据库于 1995 年由 Kanehisa Laboratories 推出 0.1 版,目前发展为一个综合性数据库,其中最核心的为 KEGG PATHWAY 和 KEGG ORTHOLOGY 数据库。在 KEGG ORTHOLOGY 数据库中,将行使相同功能的基因聚在一起,称为 Ortholog Groups (KO entries),每个 KO 包含多个基因信息,并在一至多个 pathway 中发挥作用。而在 KEGG PATHWAY 数据库中,将生物代谢通路划分为 6 类,分别为:细胞过程(Cellular Processes)、环境信息处理(Environmental Information Processing)、遗传信息处理(Genetic Information Processing)、人类疾病(Human Diseases)、新陈代谢(Metabolism)、生物体系统(Organismal Systems),其中每类又被系统分类为二、三、四层。第二层目前包括有 57个种子 pathway;第三层即为其代谢通路图;第四层为每个代谢通路图的具体注释信息。
使用KEGG注释软件KofamScan和KOfam数据库对prokka预测基因的蛋白序列进行KEGG注释。其中一个bin的KEGG pathway level2水平的CDS数量如图3-15所示,其它bins请查看结果文件夹。
图3-15 KEGG pathway level2水平CDS数量可视化
横坐标是KEGG level2水平CDS数量;纵坐标是KEGG level 2名称;颜色用于区分KEGG level 1的类型。
KEGG数据库有比较详细的通路图,我们将每个bin注释到的KO(KEGG Ortholog Groups)在通路图中高亮显示。其中一个bin的其中一个通路如图3-16所示,其它结果请看结果文件夹。

图3-16 bin 注释到的基因通路图
高亮显示的基因是在该bin中注释到的基因。对应的KO ID可以在相应的网页中查看(打开map*.html,鼠标悬浮在色块上即可)
KEGG通路的符号图例如下:

05-Bin_Taxonomy bins物种注释和进化分析
05-Bin_Taxonomy/ [bins物种注释和进化分析结果文件夹] ├── bin_abundance_taxonomy.xls [bins丰度表,同时在最后一列加上了bin的物种分类注释] ├── bin_tree.tre [bins 进化树,nwk格式] ├── phylogenetic_tree_heatmap_ID.pdf [进化树可视化,标注bin ID] ├── phylogenetic_tree_heatmap.pdf [进化树可视化,标注bin 所属的属水平分类] └── phylogenetic_tree_table.xls [与进化树中的热图对应的丰度表]
PhyloPhlAn是发表在Nature Communications上的一个用于分析微生物之间进化关系的软件[28],PhyloPhlAn3在PhyloPhlAn基础上作了很多改进,再次发表于Nature Communications[19]。PhyloPhlAn3能通过分析400多种通用微生物蛋白展示微生物(bins)之间的进化关系。同时能通过将bin与MetaRefSGB数据库[4]比对,以获取bin的物种注释信息。
图3-17是Bin之间的进化关系树状图,图中的热图展示的是bin丰度信息,树状图由R语言ggplot2和ggtree包绘制
图3-17 bins 之间的进化树
树状图中距离越近的bin,进化关系也越亲近;用不同颜色标注出了进化树中的不同微生物门水平分类;右侧热图的颜色与bin在各样本中的丰度对应。
06-Bin_Circos bins基因组Circos图
Circos是由加拿大生物信息学科学家Martin Krzywinski利用Perl语言开发的一款可以用于描述关系型数据和多维数据的基因组圈图可视化软件。2009年Circos发表在Genome research。Circos不仅能将一个物种的整个基因组呈现在一张图片中,还可以给基因组添加丰富的注释信息,如功能注释信息,差异统计信息等等。
利用每个Bin(基于contig)的Prokka蛋白预测信息,功能区注释信息(含正负链、CDS、RNA类型等信息),CAZy数据库注释结果(GH、GT、CE、PL、CBM、AA),以及GC content、GC skew的统计结果绘制Circos圈图。图3-18是其中一个bin的基因组Circos图,其它bin请查看结果文件夹。

图3-18 Bin基因组圈图
由于图片篇幅限制,图中只展示了长度最长的20条contigs。从外到内,第一圈表示属于该bin的contigs,长度用刻度表示,单位为bp;第二圈用不同颜色区分contigs上的正链和负链;第三圈用不同颜色的三角标出tRNA, rRNA, tmRNA以及CDS(编码蛋白)的编码区;第四圈标注CDS注释到CAZy数据库的碳水化合物活性酶分类,如果没有注释到任何基因,该圈可能为空;第五圈标注contigs分段(每1kb)GC含量,其中用不同颜色区分大于和小于所有contigs分段GC含量总平均值;第六圈标注contigs 分段(每1kb)GC skew值,大于和小于0用不同颜色区分。
四 常见问题解答
1. 为什么大多数bins注释不到种水平?
2. 宏基因组分析得到的优势菌为何无法通过binning得到其基因组?
五 参考文献
- Metagenomic discovery of biomass-degrading genes and genomes from cow rumen. Science 2011
- MetaBAT 2: an adaptive binning algorithm for robust and efficient genome reconstruction from metagenome assemblies. PeerJ. 2019
- AMBER: Assessment of Metagenome BinnERs. Gigascience. 2018
- Extensive Unexplored Human Microbiome Diversity Revealed by Over 150,000 Genomes from Metagenomes Spanning Age, Geography, and Lifestyle. Cell. 2019
- Compendium of 4,941 rumen metagenome-assembled genomes for rumen microbiome biology and enzyme discovery. Nature Biotechnology 2019
- Assignment of virus and antimicrobial resistance genes to microbial hosts in a complex microbial community by combined long-read assembly and proximity ligation. Genome Biol. 2019
- Quality control and preprocessing of metagenomic datasets. Bioinformatics 2011
- Cutadapt Removes Adapter Sequences From High-Throughput Sequencing Reads. Bioinformatics 2011
- Fast gapped-read alignment with Bowtie 2. Nature Methods. 2012
- The Sequence Alignment/Map format and SAMtools. Bioinformatics. 2009
- Parks DH et al. 2017. Recovery of nearly 8,000 metagenome-assembled genomes substantially expands the tree of life. Nat Microbiol 2: 1533-1542.
- CheckM: assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes. Genome Res. 2015
- dRep: a tool for fast and accurate genomic comparisons that enables improved genome recovery from metagenomes through de-replication. ISME J. 2017;11:2864–8.
- Blobology: exploring raw genome data for contaminants, symbionts and parasites using taxon-annotated GC-coverage plots. Front Genet. 2013
- Prokka: rapid prokaryotic genome annotation. Bioinformatics. 2014
- KofamKOALA: KEGG ortholog assignment based on profile HMM and adaptive score threshold. Bioinformatics. 2019 Nov 19. pii: btz859.
- Fast genome-wide functional annotation through orthology assignment by eggNOG-mapper. Submitted (2016).
- Fast and sensitive protein alignment using DIAMOND. Nat Methods. 2015
- Precise phylogenetic analysis of microbial isolates and genomes from metagenomes using PhyloPhlAn 3.0
- Trimmomatic: A flexible trimmer for Illumina Sequence Data. Bioinformatics 2014
- Metagenomic biomarker discovery and explanation. Genome Biol.12,R60 .
- eggNOG 5.0: a hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses. Nucleic Acids Res. 2019
- The Carbohydrate-Active EnZymes database (CAZy): an expert resource for glycogenomics. Nucleic acids research, 2009
- KofamKOALA: KEGG ortholog assignment based on profile HMM and adaptive score threshold. Bioinformatics. 2019
- Circos: An information aesthetic for comparative genomics. Genome Research 2009
- KEGG: new perspectives on genomes, pathways, diseases and drugs. Nucleic Acids Res. 2017
- The COG database: a tool for genome-scale analysis of protein functions and evolution. Nucleic Acids Res. 2000
- PhyloPhlAn is a new method for improved phylogenetic and taxonomic placement of microbes. Nature Communications 2013