03.Gene_predict/
├──all_sample.prokka.stat.txt [所有样本prokka注释结果]
├──all_ncRNA.stat.xls [ncRNA结果统计表]
├── bacant
│ └── *.bacant
│ ├── *.AMR.possible.tsv【耐药基因可能结果】
│ ├── *.AMR.tsv【耐药基因最优结果】
│ ├── *.integrons.detail.tsv【整合子基因结果】
│ ├──*.replicon.tsv【复制子结果】
│ ├── *.transposon.filter.tsv【转座子最优结果】
│ └── *.transposon.possible.tsv【转座子可能结果】
├── COG
│ ├── *.cog.txt [COG比对结果]
│ └── plot [COG图片]
│ ├── cog_summed_up_summary_cog_color.pdf[COG汇总图片]
│ ├── cog_summed_up_summary_cog_color.png[COG汇总图片]
│ ├── cog_summed_up_summary_ggplot2_color.pdf[COG汇总图片]
│ └── cog_summed_up_summary_ggplot2_color.png[COG汇总图片]
│ │ ├── *.cog.anno.tsv[COG结果文档]
│ │ ├── *.cog.catalog.tsv[COG结果文档,详细说明参见KEGG]
│ │ └── *.cog.txt[COG结果文档]
├── EC_number
│ └── *.ec_number.txt [ec_number结果]
├── ncRNA
│ ├── *.denovo.rRNA.fa [rRNA预测结果,fasta格式]
│ ├── *.ncRNA.stat.xls [ncRNA 预测结果统计表]
│ ├── *.rRNA.gff [rRNA预测结果,gff格式]
│ ├── *.srna.gff [sRNA预测结果,gff格式]
│ ├── *.tRNA.gff3[tRNA预测结果,gff格式]
│ └── *.tRNA.structure[tRNA结构预测结果]
└── prokka
├── *.faa[prokka预测的蛋白氨基酸序列]
├── *.ffn[prokka注释的核苷酸序列]
├── *.fna[用于提交的Contig序列(核苷酸)]
├── *.fsa【用于提交的Contig序列(核苷酸)]
├── *.gbk[genbank格式的注释文件]
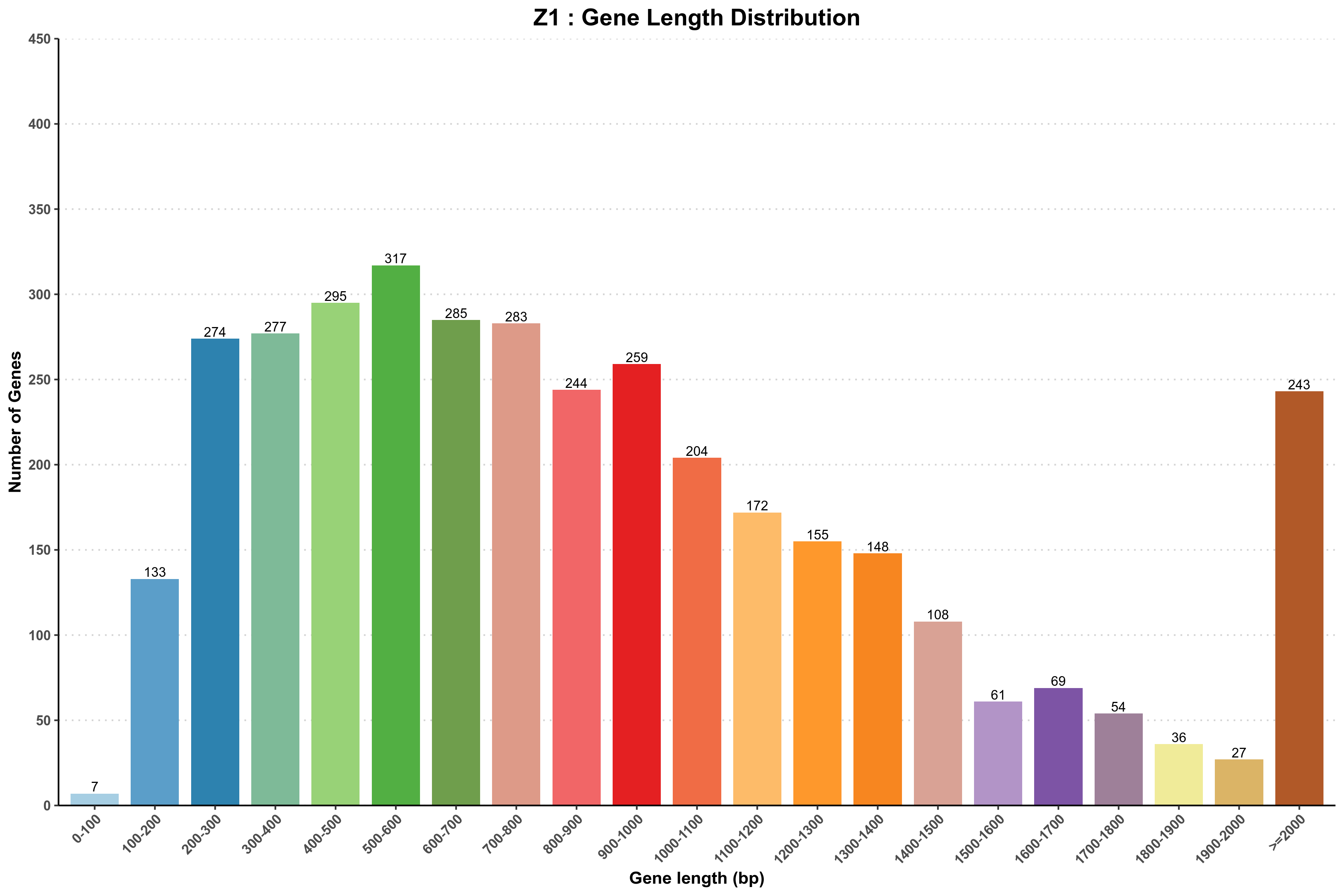
├── *_gene_length.png[基因长度统计图]
├── *.gff[包含序列和注释的GFF文件]
├── *.pdf [注释汇总统计图]
├── *.png[注释汇总统计图]
├── *.sqn[用于提交的Sequin可编辑文件]
├── *_summarize.txt[注释汇总统计,用于绘图]
├── *.tbl[用于提交的特征表(Feature table)]
├── *.tsv[注释基因列表]
└── *.txt[注释汇总统计]
使用细菌基因组功能注释工具prokka,对组装生成的assembly.fasta基因组序列文件,做基因预测和功能注释,然后用R语言分别对不同的功能注释做统计。

说明:横坐标为基因长度,纵坐标为相应基因个数
| ContigID | NumOf_ftype | NumOf_CDS | NumOf_repeat_region | NumOf_rRNA | NumOf_tmRNA | NumOf_tRNA | NumOf_gene | NumOf_COG | NumOf_EC_number |
|---|---|---|---|---|---|---|---|---|---|
| C1 | 4626 | 4542 | 1.0 | 3 | 1 | 79 | 3460 | 2824 | 1639 |
| C2 | 4626 | 4542 | 1.0 | 3 | 1 | 79 | 3460 | 2824 | 1639 |
| Z1 | 3736 | 3651 | NaN | 5 | 1 | 79 | 2456 | 2022 | 1217 |
COG,即Clusters of Orthologous Groups of proteins(直系同源蛋白簇)。COG是由NCBI创建并维护的蛋白数据库,根据细菌、藻类和真核生物完整基因组的编码蛋白系统进化关系分类构建而成。每一簇COG由直系同源蛋白序列构成,通过比对可以将某个蛋白序列归到某一个已知功能的COG中,从而可以推测该未知功能的蛋白的功能信息。 我们对基因组prokka注释结果中的.tsv文件,提取COG注释信息,然后用R语言做分类统计。
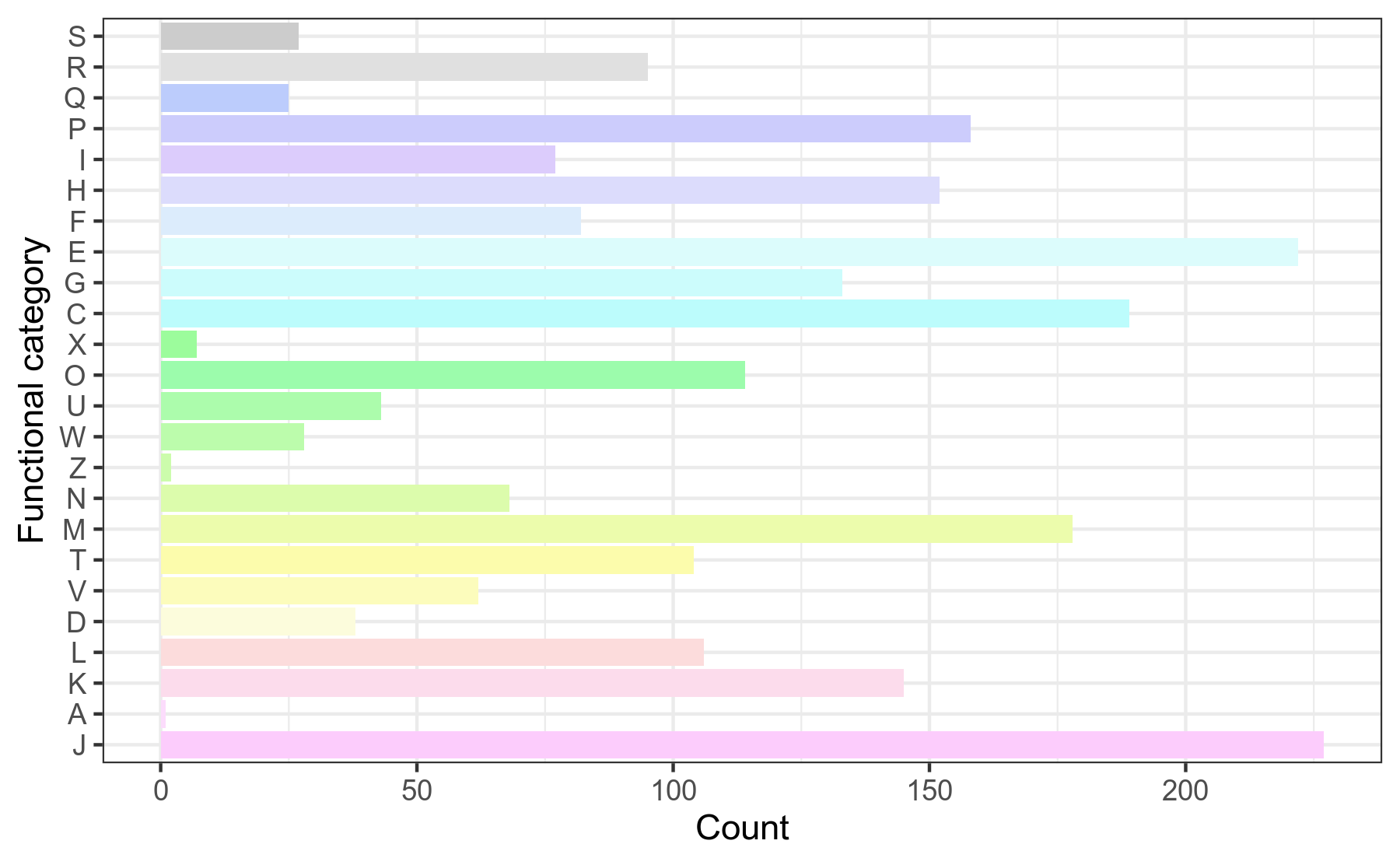
说明:横坐标为相应基因个数,纵坐标为COG分类
COG分类字母代表含义见COG分类结果
EC_number是酶学委员会(Enzyme Commission)为酶所设计的一套编号分类法,每一个酶的编号都以字母“EC”开头,接着以四段号码来表示,这些号码表示对酶作出不同层级的分类。例如三肽胺基-蛋白酶的编号为EC3.4.11.4,当中的“EC3”是指水解酶(即需要用水来将其它分子分解的酶);“EC3.4”是指与肽键作用的水解酶;“EC3.4.11”是指从多肽中分开胺基末端的水解酶;“EC3.4.11.4”则是指从三肽中分开胺基末端的水解酶。 我们对基因组prokka注释结果中的.tsv文件,提取EC_number注释信息,整理成单独的表格文件.
非编码RNA(ncRNA)是一类执行多种生物学功能的RNA分子,其本身并不携带翻译为蛋白质的信息,直接在RNA水平对生命活动发挥作用。对于微生物而言,研究较为普遍的包括sRNA、rRNA、tRNA。
tRNA:转运RNA(Transfer RNA),又称传送核糖核酸、转移核糖核酸,通常简称为tRNA,是一种由76-90个核苷酸所组成的RNA,其3'端可以在氨酰-tRNA合成酶催化之下,接附特定种类的氨基酸。转译的过程中,tRNA可借由自身的反密码子识别mRNA上的密码子,将该密码子对应的氨基酸转运至核糖体合成中的多肽链上。本分析中通过tRNAscan-SE软件对tRNA进行预测。
rRNA:即核糖体RNA,rRNA在相邻物种中高度保守。rRNA的预测方法有两种,一是通过与近缘参考序列的rRNA库比对找到rRNA,二是用rRNAmmer软件预测rRNA。
sRNA:小RNA,首先进行Rfam database比对注释,接着用cmsearch程序(参数默认)确定最终的sRNA。
snRNA:(small nuclearRNA,小核RNA),它是真核生物转录后加工过程中RNA剪接体(spilceosome)的主要成分。
miRNA:MicroRNA(miRNA)是在真核生物中发现的一类内源性的具有调控功能的非编码RNA,前体全长约90bp,其成熟miRNA大小长约20~25个核苷酸(nt)。miRNA广泛存在于真核生物中,是一组不编码蛋白质的短序列RNA,它本身不具有开放阅读框(ORF),对基因的表达具有调控作用。
sRNA、snRNA、miRNA的预测原理类似,首先用Rfam软件进行Rfam database比对注释,接着用其cmsearch程序(参数默认)确定最终的sRNA、snRNA、miRNA。由于细菌是原核生物,故ncRNA 的类型主要指 tRNA、rRNA及 sRNA 三种。
此次分析中的tRNA和rRNA预测的结果可能和prokka基因预测的结果不同,这是由于采用的软件不同导致的。
详细结果说明见ncRNA注释结果
各样本ncRNA结果统计表格如下:
| sampleID | type | count | total_length | average_length |
|---|---|---|---|---|
| C1 | tRNA | 78 | 5823 | 74.65 |
| C1 | 16s_rRNA | 1 | 1529 | 1529.00 |
| C1 | 23s_rRNA | 1 | 2900 | 2900.00 |
| C1 | 5s_rRNA | 1 | 114 | 114.00 |
| C1 | sRNA | 114 | 12173 | 106.78 |
| C2 | tRNA | 78 | 5823 | 74.65 |
| C2 | 16s_rRNA | 1 | 1529 | 1529.00 |
| C2 | 23s_rRNA | 1 | 2900 | 2900.00 |
| C2 | 5s_rRNA | 1 | 114 | 114.00 |
| C2 | sRNA | 114 | 12173 | 106.78 |
| Z1 | tRNA | 77 | 5736 | 74.49 |
| Z1 | 16s_rRNA | 1 | 1530 | 1530.00 |
| Z1 | 23s_rRNA | 1 | 1723 | 1723.00 |
| Z1 | 5s_rRNA | 3 | 342 | 114.00 |
| Z1 | sRNA | 19 | 2255 | 118.68 |
| 软件 | 版本 |
|---|---|
| prokka | 1.14.6 |